Introduction
microRNA(miRNA)는 ~22 nucleotide(nt)의 길이를 갖는 단일 가닥 RNA 분자로서, Argonaute 단백질(AGO)과 복합체를 이뤄 target mRNA의 3’UTR에 결합하여 해당 유전자의 발현을 억제하는 미세 조절자 역할을 한다. 최소 60%에 달하는 인간 mRNA들이 miRNA의 조절을 받고 있으며, 암을 비롯한 여러 질병과도 관련이 되어있는 중요한 생체 내 조절자 역할을 수행한다.
miRNA는 target mRNA를 Watson-Crick nucleotide 결합을 통해 인식한다. 이 때, 6 nucleotides의 seed 지역이 중요하다고 알려져 있으며, 이를 바탕으로 miRNA가 target mRNA를 억제하는 4가지 유형의 표준적 결합규칙(canonical site type, CST)들이 실험을 통하여 입증되었다. 이에 더하여, AGO 교차 결합과 면역 침강을 응용한 고성능 염기서열 결정법(Argonaute crosslinking and immunoprecipitation followed by high-throughput sequencing, AGO CLIP-seq) 등의 생화학적 접근법을 통해 몇 가지 비표준 결합규칙(non-canonical site type, NST)들이 보고된 바 있다. 그러나, 이들 선행 연구들은 다음과 같은 명백한 한계가 존재한다. 먼저, AGO CLIP-seq을 이용한 연구의 경우 miRNA와 target mRNA 사이의 결합에 대한 정보는 제공하지만, 결합한다는 사실만으로는 해당 mRNA의 발현이 실제로 억제된다고 보장하기 어렵다. 또한, miRNA와 인간 mRNA들 사이에는 실로 무수히 많은 유형의 결합 형태가 일어날 수 있음에도 불구하고 선행연구들은 이들 중 극소수의 결합규칙들에 대해서만 검증을 시도했다.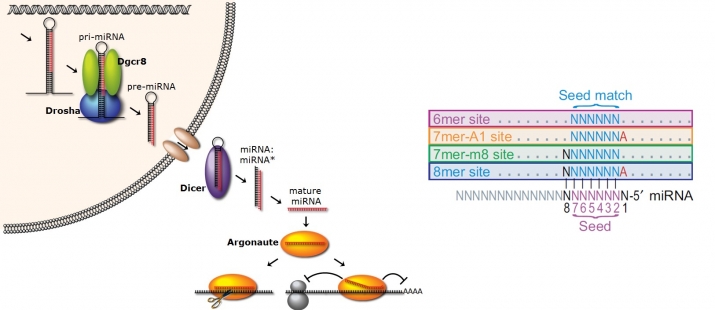
(miRNA function & canonical targeting rules (right, Baek et al. 2008))
Pipeline for discovery of functional miRNA targeting rules
miRNA와 mRNA 사이에서 일어날 수 있는 모든 결합규칙들에 대해서 해당 결합규칙이target mRNA의 발현을 억제할 수 있는지 평가하기 위해 miRNA overexpression에 대한 mRNA들의 발현 양상 변화(transcriptome response)를 측정한 microarray 데이터들을 다량 수집하고, 이를 포괄적으로 분석할 수 있는 생물정보학 분석 파이프라인을 개발했다. 20억 개에 달하는 결합규칙들 각각이 miRNA에 반응하는 mRNA들과 유의미하게 연관되어있는지 분석하기 위한 거대규모 통계학적 분석을 수행했다.
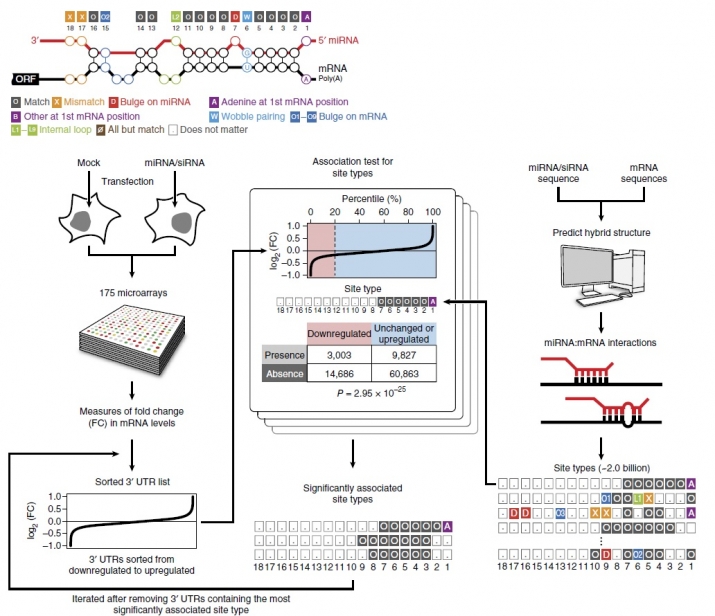
(miRNA targeting analysis pipeline (Kim, Sung, Park et al. 2016))
General rules for functional miRNA targeting
본 연구진이 개발한 생물정보학 분석 파이프라인을 이용하여 miRNA targeting에 대한 거대규모의 분석을 수행한 결과, 이미 알려진 4가지 형태의 CST들 외에 7 가지의 NST들을 추가로 발굴했으며, 이들이 실제로 target mRNA혹은 단백질을 억제함을 독립된 데이터 분석과 추가 실험을 통해 검증했다. 또한, 척추동물 유전체 상에서 상당수의 NST target site들이 진화적으로 보존되어있음을 확인했는데, 이는 이들 NST들이 생물학적으로 중요한 역할을 담당함을 암시한다. 비록 발굴된 NST들은 기존에 알려진 4가지 CST들보다 target mRNA의 발현을 억제하는 정도는 약하지만, target의 갯수가 많아 인간 유전자 발현에 미치는 영향은 CST의 그것과 필적할 정도였다. 본 연구를 통해 그 동안 전례가 없었던 포괄적인 miRNA targeting 연구를 수행함으로써, miRNA에 의한 인간 유전자 조절 네트워크가 기존에 알려진 것보다 훨씬 복잡하다는 것을 밝혀냈다 (Kim, Sung, Park et al. 2016). 본 연구에서 제시된 포괄적인 miRNA의 기능적 targeting 규칙들로 인해 miRNA에 의한 유전자 제어 기작에 대해 보다 명확히 이해할 수 있게 되었고, 본 연구실에서는 이와 관련한 후속 연구를 진행하고 있다.
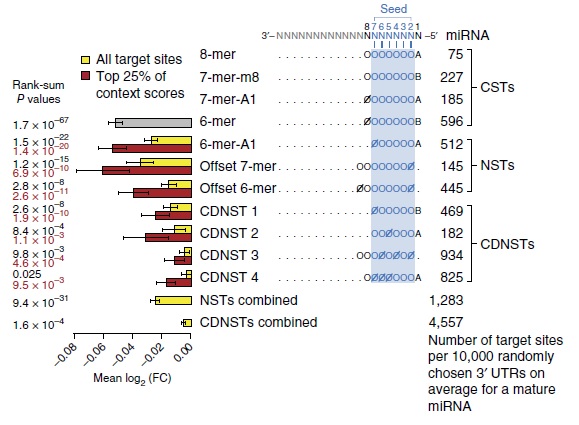
(Discovery of functional miRNA targeting rules (Kim, Sung, Park et al. 2016))
References
1. Baek, D., Villén, J., Shin, C., Camargo, F. D., Gygi, S. P., & Bartel, D. P. The impact of microRNAs on protein output. Nature (2008).
2. Kim, D., Sung, Y. M., Park, J., Kim, S., Kim, J., Park, J., ... & Baek, D. General rules for functional microRNA targeting. Nature Genetics (2016).
